Todo lo que tenés que saber sobre la reacción en cadena de la polimerasa (PCR), y no morir en el intento.
La reacción en cadena de la polimerasa (PCR por sus siglas en inglés Polymerase Chain Reaction) es una técnica que revolucionó la biología molecular, sin la cual no se podría, entre otras cosas, diagnosticar mutaciones en el ámbito de la oncología (HERB2, p53 entre muchas), la medicina forense, medicina prenatal, y la que permitió el diagnóstico temprano de múltiples infecciones, ocultas para la medicina con otras técnicas.
Haciendo un poco de historia
La PCR surgió en 1971, cuando se explica la técnica de cómo replicar un fragmento de ADN utilizando 2 iniciadores (1). Pero recién en la década del 80´ es cuando la llevaron a cabo por primera vez. En dicha oportunidad utilizaron ADN polimerasa de Escherichia Coli y los respectivos nucleótidos. Lograron replicar en forma exponencial un fragmento de ADN.
La técnica implicaba llevar la mezcla a 95º para desnaturalizar la doble hélice de ADN, calentando la mezcla de reactivos a baño María. Pero al llegar a dichas temperaturas, la ADN polimerasa se inactivaba, por lo que la técnica resultaba muy poco eficiente dado que se requería el agregado de enzima a la mezcla; ello ocurría varias veces en el proceso. Además requería de supervisión constante.
La técnica cambió radicalmente con la aparición de enzimas termoestables (2). Se logró aislar la primera polimerasa termoestable conocida llamada Taq polimerasa, producida por la bacteria Thermus aquaticus que vive en ambientes acuáticos con temperaturas cercanas a los 100 °C. Esto permitió la automatización del proceso, sin necesidad de agregar nueva enzima en cada uno de los ciclos.
En sus orígenes se utilizaban isótopos radiactivos para marcar las copias obtenidas. La cantidad de material irradiado daba la pauta de la cantidad de copias en la muestra.
Posteriormente se desarrollaron técnicas más sencillas y menos riesgosas. La verificación del resultado se transfería a una placa electroforética con gel de agarosa. En la electroforesis los fragmentos de ADN se separan según su tamaño.
Habitualmente se incluye un marcador del mismo peso molecular, para que pueda determinarse el tamaño de los fragmentos en la muestra de PCR. Una PCR que produce un fragmento de 400 pares de bases (pb) se vería así en un gel:
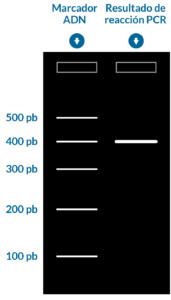
El descubrimiento de un marcador fluorescente de ADN doble cadena, el SYBR Green permitió la detección del material formado en el mismo termociclador, detectando dicha fluorescencia. Ello permite prescindir del posterior empleo de técnicas de electroforesis para la visualización de los resultados de todas las muestras.
Así, esta técnica lleva a la obtención de resultados más rápidos o con menor gasto de reactivos empleados en las técnicas de electroforesis. Si además el método se realiza en tiempo real, es decir, se observa la curva de producción de material (detectado a través de la fluorescencia), la técnica pasa de ser cualitativa a cuantitativa, dado que existe una relación directa entre la fluorescencia detectada y la cantidad de material producido, llamadose qPCR o Real time-PCR.
Principios
Para realizar la técnica se necesitan:
- Los 4 desoxirribonucleótidos trifosfato, sustratos para polimerizarnuevo ADN.
- ADN polimerasa o mezcla de distintas polimerasas con temperatura óptima alrededor de 70 °C (la más común es la Polimerasa Taq).
- ADN molde, que contiene la región de ADN que se va a amplificar.
- Termociclador, el aparato que mantiene la temperatura necesaria en cada una de las etapas que conforman un ciclo.

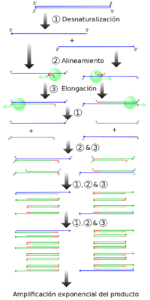
La PCR inicia con la desnaturalización o separación de la doble hélice de ADN mediante el calentamiento de la muestra a una temperatura entre 94 y 96 °C, de esta manera cada cadena queda como molde para la síntesis de una nueva cadena complementaria de ADN.
Una vez logrado ello, es necesario que los cebadores se ubiquen en los sitios específicos complementarios de las cadenas simples de la región que se va a amplificar. Para que esto suceda se baja la temperatura entre 40 y 60 °C, lo que permite la unión (alineamiento) de los iniciadores.
Finalmente, se sintetiza una nueva cadena en sentido 5’ a 3’ para lo cual se incrementa la temperatura, por lo general a 72
°C, porque es la temperatura óptima a la cual la ADN polimerasa se une a los iniciadores y comienza la replicación.
Consta de tres etapas:
- Desnaturalización
- Alineamiento
- Extensión del ADN
Estas etapas se repiten sucesivamente, y en cada nuevo ciclo se amplifica simultáneamente la región de interés de las dos cadenas complementarias. Los productos generados aumentan de manera exponencial ya que cada nueva copia sirve de molde en los ciclos posteriores, dando origen a millones de copias del fragmento seleccionado.
Rt-PCR
La reacción en cadena de la polimerasa con transcriptasa reversa (Rt-PCR) se utiliza cuando la muestra a amplificar se trata de ARN. En la RT-PCR, se retrotranscribe una hebra de ARN en ADN complementario (ADNc) usando una transcriptasa reversa, y el resultado se amplifica mediante una PCR tradicional.
Una de las características más importantes es que en el proceso de RT-PCR, el ADNc generado ya no lleva los intrones que sí tendría el ADN original. De este modo, al expresar el ADNc producto de la RT-PCR, se generará un ARNm formado exclusivamente por exones.
Esto ha permitido darle a esta técnica uno de sus usos más importantes: insertar genes eucariota en organismos procariota, fundamental para lograr que bacterias produzcan, por ejemplo, insulina humana.
La RT-qPCR permite detectar cargas virales muy bajas (20-100 copias de ARN/mL) siempre que la muestra se tome de forma adecuada y con la suficiente concentración viral y, por otra parte, es capaz de diferenciar microorganismos aunque estos tengan ciertas similitudes.
Entendiendo el CT (threshold cycle)
La interpretación del resultado de qPCR (o RT-qPCR) depende de la intensidad de la señal fluorescente en el momento que se identifica la presencia de la secuencia genética correspondiente al virus. Este trabajo lo realiza el Termociclador. Para ello, se establece una referencia, habitualmente en los primeros 10 ciclos, en el que la fluorescencia suele ser mínima.
A partir de allí, hay que calcular el umbral, entendido como el nivel de la señal fluorescente que representa un incremento estadísticamente significativo respecto al nivel basal. El umbral de ciclos o cycle threshold (Ct) es el número de ciclos en el que la señal fluorescente cruza este umbral.
Para valorar la presencia del ADN en la muestra, se determina el número de ciclos Ct de qPCR necesarios para que la prueba resulte positiva, es decir, indica el momento preciso de la amplificación en el que la prueba es capaz de identificar la presencia del fragmento investigado replicado, sin indicar la cantidad presente.
Así, el Ct es un valor semicuantitativo inversamente relacionado con la cantidad de ARN de la muestra, de manera que un número bajo de Ct está relacionado con mayor carga viral y viceversa. En una PCR estándar suelen hacerse 40 ciclos térmicos.
RT-qPCR en Sars-Cov2
RT-qPCR es el gold standard para el diagnóstico molecular de Covid-19. Ha mostrado una especificidad del 100% y una sensibilidad del 91,8%, variando entre 83% y 96,% (5).
Sensibilidad: se refiere a la cantidad mínima de ADN necesaria para que se produzca la amplificación, es decir, para obtener señal fluorescente. Se relaciona con los falsos negativos, ya que puede que una muestra sea positiva pero sea dada como negativa porque no se ha amplificado por no tener suficiente cantidad de ADN.
Especificidad: se refiere a la obtención de un solo producto amplificado. Viene determinada por los cebadores y la especificidad con la que se unen al ADN molde. De esta forma, si los cebadores tienen más de un sitio al que se pueden unir aparecerá más de un producto amplificado. Se relaciona con los falsos positivos, ya que puede que una muestra sea negativa pero sea dada como positiva porque se ha amplificado una región de ADN que no se buscaba amplificar.
Esta variabilidad se debe a distintos factores como son la precisión mostrada por el test en función del primer utilizado (téngase en cuenta que el Sars-Cov2 muestra una alta tasa de mutaciones por lo que el primer utilizado para un momento de la pandemia podría no ser adecuado para otro), el tipo y calidad de la muestra, o al tiempo transcurrido desde el inicio
de la infección y la toma de la muestra. Debe añadirse que la máxima carga viral, alcanzada antes de manifestarse los síntomas, se mantiene hasta el 3º-5º día tras la aparición de los mismos para posteriormente, ir decayendo hasta hacerse indetectable (7).
Una estrategia para minimizar errores es la posibilidad de dirigirla a identificar 2 o 3 objetivos genéticos del SARS-CoV-2, lo que evita las reacciones cruzadas con otros coronavirus endémicos y minimiza los falsos positivos al realizar la amplificación y detección de manera simultánea (8)
Valor del Ct en la predicción de la evolución en pacientes COVID-19
A pesar de que existen algunas limitaciones al interpretar el valor Ct, como serían el no expresarse con unos valores lineales, un estudio retrospectivo realizado en Nueva York mostró la relación entre el valor del Ct y la mortalidad, asociándose de forma independiente el valor Ct < 25 (en comparación con Ct > 30) tanto con la mortalidad intrahospitalaria como con la necesidad de intubación (9).
En cuanto a la capacidad de contagio, es decir, la presencia nasofaríngea o en vías aéreas inferiores, del virus por parte del paciente, el Ct mostró su utilidad, determinando incluso conductas sanitarias en la población.
Bullard et al. no consiguieron cultivar el virus con valores de Ct > 24 (muestras almacenadas a −80 °C durante 2-4 semanas), encontrando una disminución de la odds ratio del 32% por cada incremento de 1 unidad del valor Ct (10).
Otros estudios sí consiguieron cultivar el virus con valores mayores del Ct (en el 70% de los pacientes con Ct = 25; en el 20% con Ct = 30, o en el 3% con Ct = 35), mientras que otros autores refirieron una probabilidad de recuperar virus viables en el 8,3% de los pacientes con Ct > 35 cuando eran sintomáticos.
RT-qPCR para detectar variantes de interés
La técnica puede utilizarse como tamizaje de variantes de interés en evolucíon. Un ejemplo es lo realizado en San Luis por el Dr Maximiliano Juri Ayub y la Dra Jimena Manzur (UNSL). Utilizaron mutaciones presentes en la variante Omicron, que estaban presente en linajes previos no circulantes (Alpha en este caso). Es así que tomando varias muestras positivas para Sars-Covs por RT-qPCR, utilizando los primers adecuados pudieron identificar las primeras muestras de Omicron en el país, pudiéndose obviar las técnicas de genotipificación, más costosas y difíciles de acceder (12).
1. Kleppe K, Ohtsuka E, Kleppe R, Molineux I, Khorana HG. Estudios sobre polinucleótidos. XCVI. Reparar replicaciones de ADN sintético corto catalizado por ADN polimerasas. J Mol Biol. 1971 14 de marzo; 56 (2): 341-61. doi: 10.1016/0022-2836(71)90469-4. PMID: 4927950.
2. Saiki RK, Gelfand DH, Stoffel S, Scharf SJ, Higuchi R, Horn GT, Mullis KB, Erlich HA. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science. 1988 Jan 29;239(4839):487-91. doi:10.1126/science.2448875. PMID: 2448875.
3. Bustin SA, Mueller R. Real-time reverse transcription PCR (qRT-PCR) and its potential use in clinical diagnosis. Clin Sci (Lond). 2005 Oct;109(4):365-79. doi: 10.1042/CS20050086. PMID:16171460.
4. Gibson UE, Heid CA, Williams PM. A novel method for real time quantitative RT-PCR. Genome Res. 1996 Oct;6(10):995-1001. doi: 10.1101/gr.6.10.995. PMID: 8908519.
5. Bisoffi Z., Pomari E., Deiana M., Piubelli C., Ronzoni N., Beltrame A. Sensitivity, specificity and predictive values of molecular and serological tests for COVID-19 A longitudinal study in emergency room. MedRxiv. 2020 doi: 10.1101/2020.08.09.20171355.
6. Tom M., Mina J.M. To interpret the SARS-CoV-2 test, consider the cycle threshold value. Clin Infect Dis. 2020 doi: 10.1093/cid/ciaa619. ciaa619.
7. Cevik M., Tate M., Lloyd O., Maraolo A.E., Schafers J., Ho A. SARS-CoV-2 SARS-CoV, and MERS-CoV viral load dynamics, duration of viral shedding, and infectiousness: a systematic review and meta-analysis. Lancet Microbe. 2021;2:e13–e22. doi: 10.1016/S2666-5247(20)30172-5.
8. Tang Y., Schmitz J.E., Persing D.H., Strattonb C.W. Laboratory Diagnosis of COVID-19: Current Issues and Challenges. J Clin Microbiol. 2020;58:e00512–e520. doi: 10.1128/JCM.00512-20.
9. Singanayagam A., Patel M., Charlett A., Lopez Bernal J., Saliba V., Ellis J. Duration of infectiousness and correlation with RT-PCR cycle threshold values in cases of COVID-19, England, January to May 2020. Euro Surveill. 2020;25 doi: 10.2807/1560-7917.ES.2020.25.32.2001483. pii=2001483.
10. Bullard J., Dust K., Funk D., Strong J.E., Alexander D., Garnett L. Predicting infectious severe acute respiratory syndrome coronavirus 2 from diagnostic samples. Clin Infect Dis. 2020;71:2663–2666. doi: 10.1093/cid/ciaa638.
11. Jaafar R., Aherfi S., Wurtz N., Grimaldier C., Hoang V.T., Colson P. Correlation between 3790 quantitative polymerase chain reaction–positives samples and positive cell cultures, including 1941 severe acute respiratory syndrome coronavirus 2 isolates. Clin Infect Dis. 2020 doi: 10.1093/cid/ciaa1491. ciaa 1491.
12. https://universidadeshoy.com.ar/nota/72354/cientificos-puntanos-desarrollan-tecnica-para-detectar-la-omicron/
















